Metagenómica
-
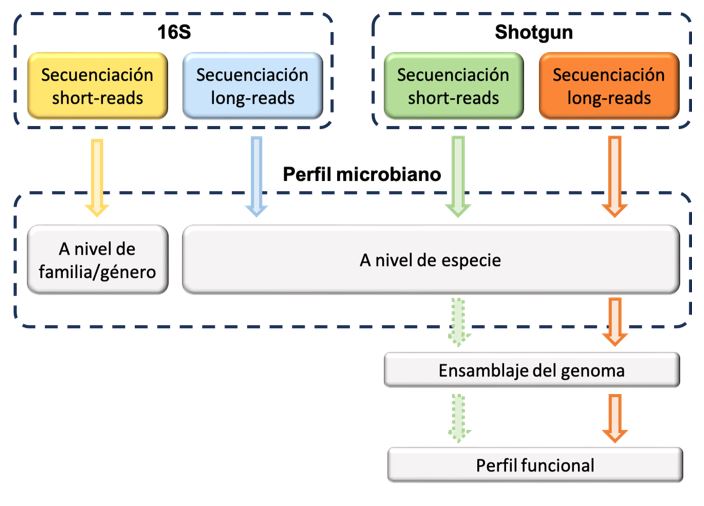
A partir de la secuenciación de las regiones 16S, 18S e ITS se obtiene información que permite diferenciar entre taxones, establecer relaciones de filogenia y clasificar organismos procariotas y eucariotas. En Seqplexing ofrecemos el análisis de los resultados de secuenciación específica de estas regiones tanto de short-reads, mediante amplicones, como de long-reads, procedentes de las plataformas Illumina, PacBIO y ONT.
-
Mediante el análisis bioinformático de los resultados obtenidos por secuenciación random Shotgun podemos detectar las bacterias presentes una muestra, así como su proporción relativa.
Además, mediante la tecnología Shotgun a partir de short-reads podemos ensamblar genomas completos procedentes de cultivos puros. En el caso de que la secuenciación se realice con long-reads se puede incluso determinar y ensamblar la secuencia completa de los diferentes organismos presentes en una única muestra. -
A partir de datos de secuenciación procedentes de metagenómica se puede obtener el perfil funcional de las especies a estudiar e identificar tanto los genes presentes, como la resistencia a antibióticos.
-
Los resultados procedentes de metatranscriptómica nos permiten detectar las rutas expresadas en los organismos presentes en una muestra.
 EN
EN

